Jetzt habe ich mal kräftig in der Buchstabensuppe herumgeführt, um ein bisschen Gefühl dafür zu bekommen, wie variabel die Sequenzen innerartlich sowie innerhalb von Gattungen ist.
Übungsopfer: diesmal der Maronenröhrling. Knapp 30 Datensätze sind als Xerocomus badius (firmiert der nicht aktuell unter Imleria?) einsortiert.
Recht große Übereinstimmung in den Sequenzen, die wenigen Abweichungen über die gesamte Sequenz verstreut, nicht selten ist der bei allen Datensätzen vorhandene Abschnitt sogar identisch.

(gelb hinterlegt sind die Abweichungen)
Systematische Abweichungen immer an bestimmten Stellen kommen nur bei 4 Proben vor, die aus Nordamerika stammen und untereinander wiederum nur minimal abweichen. Hübsch, das wirkt nachvollziehbar.
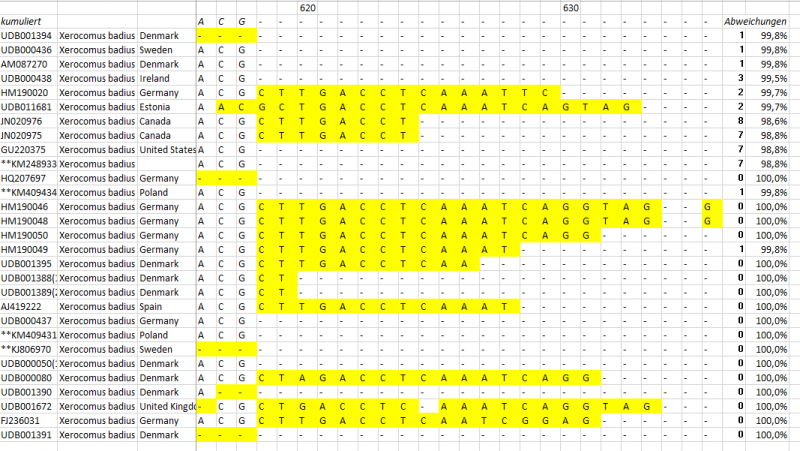
Die Abweichungen kann man sich auch noch als absolute Zahl oder prozentual ausgeben lassen, wobei ich auf den bei allen vorhandenen Abschnitt beschränkt habe, also am Anfang und am Ende ausgeklammert, was nicht bei allen Proben vorhanden war.
Als nächstes habe ich mir zwei aus der Gattung Boletus angeguckt, der die Marone ja auch mal zugeordnet war. B. edulis über 60 Datensätzen aus aller Welt. Hier erstaunliche Homogenität ohne erkennbare regionale Abweichungen.
Dann noch B. erythropus dazu, mit etwas dünnem Datenbestand, nur 7 Sequenzen aus Europa. Aber auch hier ist ein typisches Sequenzmuster deutlich erkennbar.
Diese drei also untereinandergelegt und ... Buchstabensalat. Das Alignment wird offenbar doch nicht artübergreifend gemacht und erschwert damit die Vergleichbarkeit zwischen verschiedenen Arten eher, statt sie zu erleichtert. Also habe ich es ganz entfernt und selbst versucht, ähnliche Abschnitte zu identifizieren.
Immerhin habe ich einen lange übereinstimmenden Abschnitt gefunden, über 100 Basenpaare, und dazu viele kürzere Schnipsel. Wenn ich Zeit finde, will ich auch noch Filzröhrlinge dazunehmen, in der Gattung hat die Marone ja auch mal gewohnt.

(jetzt schon mit X. submentosus - die bei allen identische Sequenz von 159 Basen, bei X. submentosus mit einer Abweichung, ist gelb hinterlegt)
Meine Fragen im Moment:
1) Wieder zurück zu den Bindestrichen. Meine Hypothese hat sich in Teilen offensichtlich als falsch erwiesen. Die sind zwar Platzhalter, wo in einer Sequenz eine Base ist und in der anderen keine, aber wenn das Alignment nur dem innerartlichen Vergleich dient und nicht dem zwischen Arten, verwundert mich immer noch oder wieder, dass häufig eine Vielzahl an Bindestrichen gesetzt wird, wo einer genügt hätte.
2) Wie funktionieren die Regeln für das Alignment genau? Wenn z.B. zwischen zwei identischen Abschnitten einmal die Basenfolge CAATGTAGC und im anderen stattdessen TTG, würde die kürzere TTG-Gruppe zerstückelt als ---T-T-G-, um die bestmögliche Übereinstimmung (3 Basen) zu erreichen, oder lässt man sie zusammen (--TTG---- oder -----TTG-, je 2 Basen oder gar TTG------ bzw. ------TTG, 0 Basen)?


